Årets Nobelpris i kemi hjälper oss att förstå livets byggstenar
Amerikanen David Baker har lyckats med konststycket att skapa helt nya proteiner – livets byggstenar som styr alla biologiska processer i kroppen. Han delar 2024 års Nobelpris i kemi med Demis Hassabis och John M. Jumper från Google DeepMind, Storbritannien, som ligger bakom AI-verktyget AlphaFold2. ”Nu kan det ta minuter i stället för år att få en bra uppfattning av hur ett protein ser ut och hur det kan fungera”, säger KI-professorn Luca Jovine som tidigt inledde ett samarbete med DeepMind.
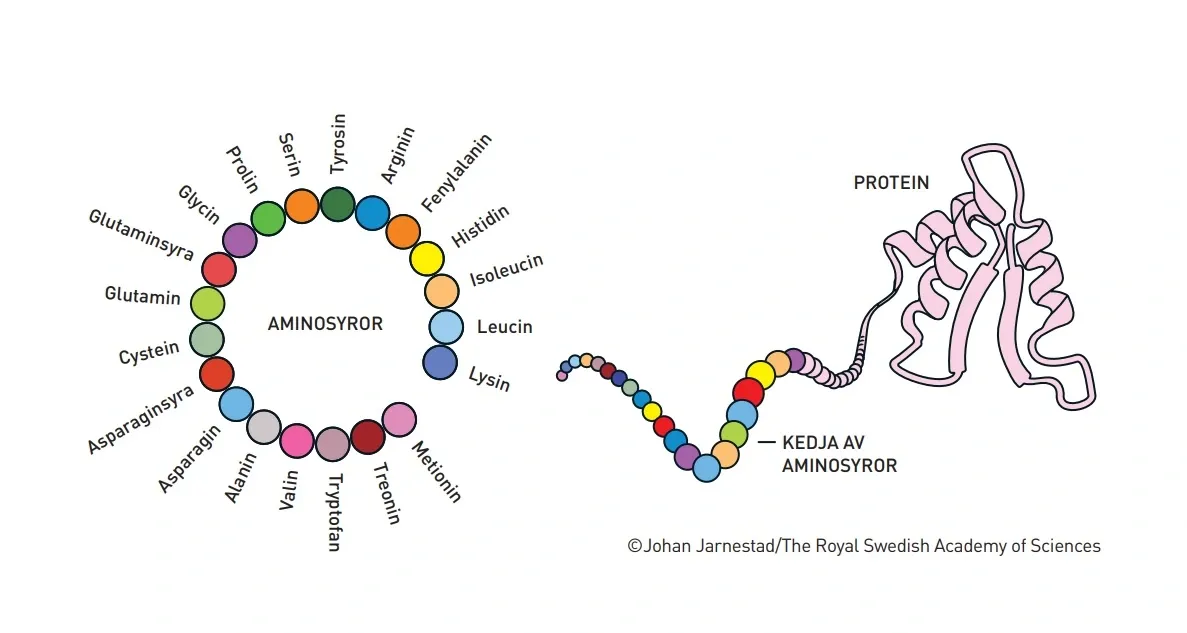
Proteiner styr alla kemiska reaktioner som ger förutsättningar för liv. I våra kroppar fungerar de till exempel som hormoner, signalämnen, antikroppar och byggstenar i olika vävnader.

Årets Nobelpris i kemi belönar två viktiga upptäckter om proteiner. Den ena handlar om konsten att bygga egna proteiner med önskade egenskaper, något som David Baker vid University of Washington, USA, lyckades med för drygt 20 år sedan. Han får ena hälften av kemipriset ”för datorbaserad proteindesign” och har skapat proteiner som kan fungera som bland annat läkemedel, vaccin, nanomaterial och sensorer.
Viktigt för medicinsk forskning
Den andra hälften delas mellan Demis Hassabis och John Jumper, Google DeepMind, ”för proteinstrukturprediktion”.

– Det handlar om att kunna förutspå hur proteiner ser ut i 3D och därmed få insikter om deras funktion och interaktioner med andra molekyler i kroppen. Det är helt avgörande för att kunna förstå biologiska processer, hur sjukdomar uppstår och hur de skulle kunna behandlas, berättar Luca Jovine, professor i strukturbiologi vid institutionen för medicin, Huddinge, Karolinska Institutet.
Han studerar bland annat interaktionen mellan ägg och spermier vid befruktning och hur mutationer i ägghöljets proteiner kan orsaka kvinnlig infertilitet. Strax efter att Demis Hassabis och John Jumper presenterade AI-modellen AlphaFold2 år 2020 var Luca Jovines forskargrupp en av de första att inleda ett samarbete med DeepMind. Sedan dess har de publicerat två vetenskapliga artiklar tillsammans med bland andra John Jumper.
Demokratisering av AI-verktyg
– Det har varit ett nöje att samarbeta med John, som är en väldigt lättsam, trevlig och jordnära person. Att han och Demis Hassabis får Nobelpriset i kemi är både väntat och välförtjänt. Jag tycker också att det är bra att priset delas med David Baker, vars Rosetta-programsvit var avgörande inte bara för framgången med ny proteindesign utan också för att driva på proteinstrukturprediktion. Koderna för både Rosetta och AlphaFold2 har gjorts öppet tillgängliga, vilket har varit viktigt för demokratiseringen av dessa kraftfulla AI-verktyg liksom för utvecklingen av andra verktyg som bygger på dem.
Proteiner består av aminosyror som är sammanlänkade i långa kedjor och som veckas till specifika tredimensionella strukturer som är avgörande för deras olika funktioner. AlphaFold2 kan förutsäga strukturen hos miljontals proteiner baserat på aminosyrornas sekvens.
– Även om det inte är perfekt är träffsäkerheten generellt sett mycket hög, vilket gör det till ett otroligt värdefullt verktyg för alla forskare som studerar proteiner. Det har tagit den biologiska och medicinska forskningen med storm eftersom vi nu i många fall kan få fram en högkvalitativ modell av ett proteins struktur på några minuter i stället för år, säger Luca Jovine.
Ett bra komplement
Han betonar dock att det fortfarande finns ett stort behov av traditionella metoder som gör det möjligt att experimentellt bestämma proteiners struktur. De ger en högre detaljnivå och kan hantera strukturen hos mycket stora makromolekylära komplex. När AlphaFold2 först presenterades såg en del forskare den som ett hot mot traditionella metoder som röntgenkristallografi, kryo-EM och NMR, men Luca Jovine såg den genast som en stor tillgång och ett komplement.
– Genom att kombinera traditionella metoder med AI kan vi snabba upp processen och få fram mycket mer information. Denna kunskap kan sedan användas för att utveckla nya läkemedel eller för att förutsäga om en mutation i ett protein sannolikt kommer att leda till sjukdom eller inte. Det här är grundläggande frågor som många medicinska forskare arbetar med och vars kliniska relevans gör att den långsiktiga potentialen för AI-verktyg är mycket stor.
Läkemedel för psykisk sjukdom

Per Svenningsson, överläkare och professor i neurologi vid institutionen för klinisk neurovetenskap, Karolinska Institutet, har använt AlphaFold2 för att identifiera läkemedelskandidater för psykiska sjukdomar som schizofreni och psykos.
– Det hjälper oss att designa nya läkemedelsmolekyler på ett effektivt sätt och är ett bra komplement till experimentella metoder, säger han.
Forskargruppen använder tekniken för att bestämma den tredimensionella strukturen av så kallade G-protein kopplade receptorer, en typ av proteiner i kroppen som är vanliga måltavlor för läkemedel. De kan då få en uppfattning av hur effektivt olika molekyler binder till dessa måltavlor och därmed vilken potential de har att utvecklas till nya läkemedel.

– Vi har kunnat designa flera extremt potenta läkemedelskandidater för ett målprotein som vi inte hade några tidigare strukturella data om – något som hade varit ouppnåeligt för inte så länge sedan, säger Marcus Saarinen, doktorand i Per Svenningssons forskargrupp.
På senare tid har de även kunnat använda AI-tekniken för att utforska större proteinkomplex. Det är centralt för att förstå hur måltavlorna fungerar som en del av ett större system.
– Tekniken förbättras kontinuerligt och möjligheterna inom syntetisk biologi och läkemedelsutveckling är oerhört spännande, avslutar Marcus Saarinen.
Text: Felicia Lindberg
