Kristallografi, cryo-EM och AlphaFold: en perfekt trio som kastar ljus över viktiga antibakteriella proteiner
Ett team, under ledning av Karolinska Institutet, har kombinerat artificiell intelligens (AI) och strukturbiologi för att få insikter om två liknande protein som kan förhindra bakteriell infektion i urinvägarna respektive mag-tarmsystemet. Resultatet har nu publicerats i "Nature Structural & Molecular Biology".
"Dessa fynd skulle i princip kunna utforskas för att utveckla ett lockbetesalternativ till konventionella antibiotikabehandlingar. Och vårt strukturella arbete förklarar många av de mutationer associerade med njursjukdomar, samt ger information om en proteinregion vars interaktion med filtrerade lätta kedjor är inblandade i så kallad "cast nephropathy", säger professor Luca Jovine vid Institutionen för biovetenskaper och näringslära och huvudförfattare av artikeln.
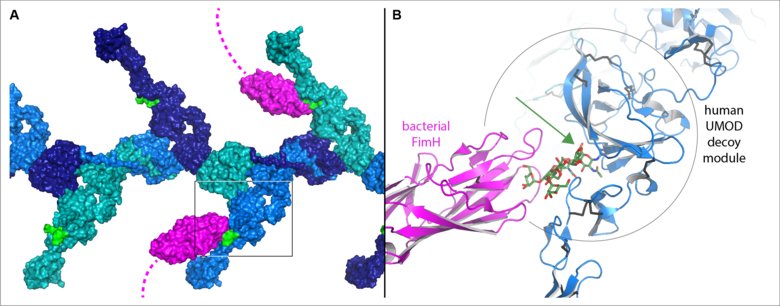
Uromodulin (UMOD) är ett protein vars fibrer spelar en viktig roll för att förebygga urinvägsinfektion, som är den vanligaste formen av icke-epidemisk bakterieinfektion och drabbar cirka 150 miljoner människor per år. UMOD bär en speciell sockerkedja som inte modifieras som andra och som förblir av högmannostyp. Denna fungerar som ett lockbete genom att efterlikna andra sockerkedjor med hög mannoshalt på ytan av urinvägarna, till vilka bakterier normalt skulle fästa med hjälp av ett sockerbindande protein som kallas FimH i spetsen av deras hårliknande bihang. I stället binder bakterierna till sockerkedjan i UMOD och avlägsnas så småningom genom urinen. Under de senaste åren har bevis visat att glykoprotein 2 (GP2), en UMOD-liknande molekyl som produceras i bukspottkörteln och tarmen, spelar en liknande roll för att motverka bakteriella infektioner i mag-tarmsystemet..
"Vi ville ta reda på vilken sockerkedja i GP2 som motsvarade högmannoskedjan i UMOD och förstå hur proteinerna lyckas bibehålla dessa sockerarter som 'speciella', så att de fungerar som ett lockbete för bakterier", förklarar Luca Jovine.
AI positionerar sig som ett verktyg inom strukturbiologi
I samarbete med Daniele de Sanctis på ESRF synchrotron in Grenoble och Marta Carroni på SciLifeLab i Stockholm, lyckades teamet samla in röntgenkristallografisk och kryo-elektronmikroskopisk (EM) data av GP2 respektive UMOD. Men olika tekniska problem komplicerade tolkningen. Det var vid denna tidpunkt som forskarna inledde ett samarbete med DeepMind och teamet bakom AlphaFold - ett maskininlärningsprogram som väckte stor uppmärksamhet inom strukturbiologi-sfären baserat på dess prestation i tävlingen "the 14th Critical Assessment of Structural Prediction". Det visade sig att även om AlphaFold aldrig hade "sett" UMOD eller GP2 tidigare, var dess förutsägelser extremt bra. Detta gjorde så att teamet, som också inkluderade Bin Wu vid Nanyang Technological University i Singapore och Nao Yamakawa vid Lille University, både kunde tolka röntgendatan av GP2 och få ihop lågupplösta kryo-EM-kartor över UMOD.
Arbetet avslöjade att den mest funktionella lockbetsmodulen för UMOD och GP2 bestod av två delar åtskilda av en springa. Även om sockerkedjorna som band bakterierna fästs vid olika områden av GP2- och UMOD-sekvenserna, visar 3D-strukturerna att deras bas i båda fallen är placerad i denna springa. Detta skulle förklara hur dessa specifika sockerkedjor skyddas från modifiering och hur de därmed kan presenteras som krokar som fiskar efter patogena bakterier (Figur 1).
